Detección y caracterización molecular de Cryptosporidium spp. en terneros de lechería de la provincia de Valdivia, Chile
Detection and molecular characterization of Cryptosporidium spp. in dairy calves from the Valdivia province, Chile

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-CompartirIgual 4.0.
Mostrar biografía de los autores
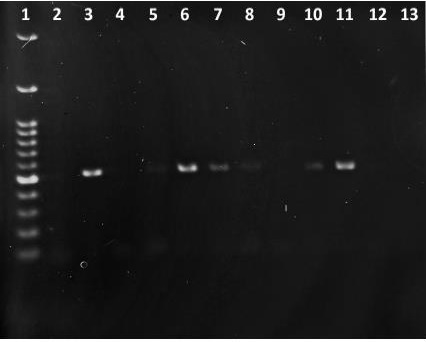
Objetivo: Detectar ooquistes ácido-alcohol resistentes de Cryptosporidium spp. en terneros mediante tinción Ziehl-Neelsen (ZN) y caracterizar molecularmente las especies y subtipos presentes en la provincia de Valdivia, a través del uso de herramientas moleculares con los genes 18S rARN y gp60 que codifica una glicoproteína de 60 kDa. Materiales y métodos: 275 muestras de heces de terneros pertenecientes a 15 predios lecheros de la provincia de Valdivia, fueron analizadas mediante tinción de ZN. Las muestras positivas a ZN, se analizaron por PCR mediante la amplificación de una región de los genes 18S rARN y gp60, con posterior secuenciación. Resultados: 30 muestras resultaron positivas a ZN, de las cuales el 46.6% (14/30) logró ser confirmado mediante PCR para 18S rARN y de estas, el 42.8% (6/14) fueron positivas a la PCR para gp60. La secuenciación arrojó la presencia de C. parvum en el 100% de las muestras y los subtipos IIaA15G2R1 (3/6), IIaA14G1R1 (2/6) y IIaA17G1R1 (1/6). Conclusiones: El subtipo IIaA15G2R1 predominó, encontrándose en dos comunas de la provincia. El subtipo IIaA14G1R1 fue encontrado en dos predios de una misma comuna y su hallazgo es el primero en bovinos en Chile y el continente. Finalmente, el subtipo IIaA17G2R1 ha sido encontrado esporádicamente en terneros, al igual que en esta investigación. Las tres variantes han sido reportadas en humanos, sin embargo, no hay registros de estudios que caractericen los subtipos de C. parvum en sistemas de crianza de terneros y trabajadores simultáneamente en Chile, por consiguiente, se debe indagar en el tema.
Visitas del artículo 860 | Visitas PDF























